
Informações
Curriculo...
Informações para Candidatos à Orientação
3.2 Mapas, Atlas e Sistemas de Referência *
Atlas Probabilísticos *
Atlas Baseado em Densidade (Intensidade) *
Atlas Baseado em Deformação *
Atlas Baseado em Rótulos *
3.4
Ferramentas para Análise e Preprocessamento, Segmentação
e Parcelamento *
Atlas cerebrais são mapas do cérebro referenciados em um sistema de coordenadas esterotáxicas [8], coordenadas, estas, que são baseadas em marcos anatômicos e, por essa razão, não são absolutas. E com a grande variabilidade neuroanatômica [2,17] é necessário ajustar a imagem do cérebro do paciente a do Atlas, seja por transformações geométricas aliadas a análise probabilística, ou por deformações aplicadas a imagem; para que se possam fazer comparações [8,2,4].
Segundo Toga et. al. a maior parte dos Atlas são baseados na anatomia de um único paciente em um espaço estereotáxico. Mapas de diversas modalidades podem ser comparados com estes Atlas, e descobrir-se regiões de ativação e outras coisas. Os primeiros Atlas eram baseados em crio-secções de um, ou poucos, indivíduos post-morten; Dentre os diversos Atlas feitos, os mais usados são o de Talairach e Tournoux (1988) e de Schaltenbrand e Wahren (1977)[2].
Meios de criar uma visualização volumétrica a partir de uma seqüência de imagens incluem a renderização baseada em superfícies, extraindo-se informação do contorno das imagens, e a renderização baseada em voxel, que interpreta o espaço entre as imagens na seqüência como a terceira dimensão do pixel, criando um voxel (um pixel volumétrico) que mantém as informações de tons de cinza das imagens originais [8]. Estes dados dos tons de cinza contêm informações sobre os tecidos, e seu significado dependerá da modalidade da aquisição da imagem.
Além disto, há ferramentas que se especializam em mapear estruturas específicas do cérebro, mas que mantém certo grau de variabilidade. Este é o caso do corpo caloso, do hipocampo e outras estruturas [10,11].
Uma ferramenta, o Mindboggle[12], apenas faz rotulação de áreas do cérebro do paciente comparando através de uma técnica de "espalhamento cerebral" (scatterbrained) a outros Atlas.
A variedade neuroanatômica é ainda mais marcante quando se refere ao córtex cerebral humano. Este tem uma estrutura muito complexa, o que pode sugerir que suas estruturas tenham auto-similaridade. Isto é, uma natureza fractal. A ontogênese do cérebro humano é influenciada por fatores genéticos, mas também por influências externas [1,13] que favorecem a variabilidade entre espécimes e contribuem para a grande complexidade deste órgão. Segundo Kiselev et. al, [3] se o córtex cerebral humano mostrar-se ser uma estrutura fractal, anomalias no padrão de giros, que são difíceis de se avaliar com técnicas morfométricas, poderiam ser estudadas com análise fractal. Vários trabalhos apontam para essa possibilidade [3,4,5,6], e associam algumas doenças a anormalidades na dimensão fractal como no caso de Free et. al [6], e Thompson, et.al [5].
3.2 Mapas, Atlas e Sistemas de Referência
Um mapa cerebral é a representação gráfica de uma modalidade de aquisição de imagens do cérebro. Foram criadas diversas formas de enxergar o cérebro, como Tomografias Computadorizadas Tridimensionais, Espécimes Anatômicos, meios de visualizar cito-arquitetura de regiões, áreas de relações químicas, MRI, PET, entre outros [2].
Os primeiros Atlas cerebrais eram baseados no estudo anatômico de indivíduos post-mortem. Alguns Atlas mais recentes de referência anatômica baseados em crio-secções do cérebro podem ter resoluções espaciais chegando a 100 microns/voxel. Com isso muitas estruturas podem ser visualizadas.
Atlas baseados em MRI permitiram o estudo de cérebros de indivíduos in-vivo, mas perdem em termos de resolução.
Também há Atlas que visam mapear modalidades adquiridas de diversos sensores (PET, CT, MRI) num mesmo sistema de coordenadas tridimensionais. A estes Atlas, dá-se o nome de Atlas Multi-Modais[2].
Um Atlas cerebral seria então um conjunto destes mapas do cérebro referenciados em um sistema de coordenadas esterotáxicas [8]. Estas são coordenadas usadas na localização de estruturas cerebrais de forma confiável entre diferentes indivíduos.
Estas coordenadas são normalizadas, para que usando transformações (rotação, translação e escalonamento), o cérebro do paciente possa ser encaixado nas coordenadas do Atlas em questão. Essas transformações são baseadas em marcos anatômicos como a linha formada pela Comissura Anterior e Posterior no caso do Atlas de Talairach. Estas transformações visam criar um sistema de coordenadas esterotáxicas padrão para que haja uma relação entre a localização de estruturas no cérebro do Atlas e a localização de estruturas no cérebro do paciente. Segundo Toga et. al. [8] vários estudos foram feitos examinando a variabilidade entre estruturas que ocorre no uso de sistemas de coordenadas, mas nenhum produziu alternativas universalmente aceitáveis.
Sistema de Coordenadas de Talairach é baseado em um cérebro de uma judia, alcoólatra, de 60 anos de idade; certamente fora dos padrões médios do cérebro jovem. Como foi desenvolvido com a idéia de planejamento cirúrgico para cirurgias em estruturas profundas no cérebro, não leva em consideração a distinção entre hemisférios. Além de ter cortes com espaçamento superior a 4mm. No entanto este sistema de coordenadas é o padrão, pois proporciona um framework para análise automática e tridimensional entre indivíduos distintos.
O Atlas de Schaltenbrand e Wahren possui cortes que variam de 1 a 4 mm de espessura [19] de um cérebro marcado para evidenciar regiões talâmicas. Também é baseado na linha formada entre a Comissura Anterior e Posterior.
Há outros exemplos de Sistemas de Referência como o proposto por Rehm et. al [21] que se baseia em pontos anatômicos no cerebelo. Este sistema de coordenadas visava contornar problemas de extrema variabilidade usando-se o sistema de coordenadas de Talairach, quando o cerebelo era o foco da pesquisa.
Outro sistema de coordenada propõe [22] um mapeamento bidimensional do córtex tratando-o como uma superfície em uma esfera. Os padrões de dobra do córtex são então mapeados a esta superfície. O artigo de Fischl et. al.[22] compara este método ao de Talairach, e aponta uma menor variabilidade quando se é feita a média de vários indivíduos com a técnica proposta.
Como visto em Toga, et. al. [2] o uso de esquemas de normalização espacial baseados em características da substância branca tem ainda que acomodar completamente a estrutura mais variável do cérebro, o córtex. O córtex é também uma área de interesse para a maioria dos estudos de ativação de função. Variações normais consideráveis na geometria dos sulcos têm sido encontradas em várias localidades no córtex cerebral. Análises recentes de tridimensionalidade encontraram padrões bastante heterogêneos de variabilidade anatômica entre cérebros de indivíduos normais e enfermos, post-mortem e in-vivo.
Isto também implica que um Atlas de um cérebro padrão dificilmente atenderá a todas as variações entre as pessoas, sendo impossível criar uma representação fiel a todos os cérebros [14,15].
Outra abordagem é focar a atenção à geometria dos sulcos e giros cerebrais, e extrair relações comparativas entre o cérebro do paciente e o cérebro do Atlas. Para este fim as imagens do Atlas ou as dos pacientes são deformadas para que tanto os marcos anatômicos, quanto os sulcos e giros coincidam nas duas séries de imagens.
Há duas abordagens para essa filosofia, pode-se tanto transformar um cérebro de um Atlas conhecido para que se encaixe no cérebro do paciente, levando consigo todas as informações do Atlas sem alterar a relação entre as estruturas cerebrais do paciente. Ou deformar a imagem do cérebro do paciente para que se encaixe na de um Atlas, tendo assim um sistema de coordenadas comuns para avaliar vários indivíduos dentro de um contexto [2]. Estas transformações requerem que qualquer parte da anatomia do cérebro presente no Atlas ou no cérebro do paciente, pequena ou não, possa girar, entortar, aumentar ou diminuir para se encaixar a outra imagem. Além disto, devem manter a topologia e a conectividade apesar destas transformações complexas.
Toga et. al [2]comenta uma abordagem para resolver esse problema usando física de materiais na deformação de imagens. Nesta abordagem, o meio é tido com uma matéria elástica ou líquido viscoso, e as forças nele submetidas se comportam como na dinâmica de materiais.
Como dito antes a variabilidade anatômica entre indivíduos é muito grande para que um Atlas possa representar fidedignamente todos os cérebros, mais especificamente os padrões de girificação do córtex cerebral[17]. Sendo assim para analisar uma população, podendo fazer-se comparações entre indivíduos é necessário que as representações dos dados individuais sejam estendidas para populações. Partindo da premissa que em certas circunstâncias um limite de confiança estatística é mais apropriado que uma representação absoluta da neuroanatomia.[2]
Os métodos de criação de Atlas cerebrais probabilísticos geralmente caem em três grandes categorias: Baseados em Densidade (Intensidade), Baseado em Deformações e Baseado em Rótulos.
Atlas Baseado em Densidade (Intensidade)
Abordagens iniciais aos Atlas
voltados a populações concentraram em gerar uma representação
"média" da anatomia tirando a média de intensidade de várias
séries MRI [2]. Um exemplo desta abordagem é o mapa do Instituto
Neurológico de Montreal (MNI em inglês), o BrainWeb, que faz
a transformação linear em cada imagem para que fiquem em
um espaço esterotáxico, normaliza pela intensidade e tira
a média voxel-por-voxel, produzindo um conjunto de dados com a média
das intensidades.
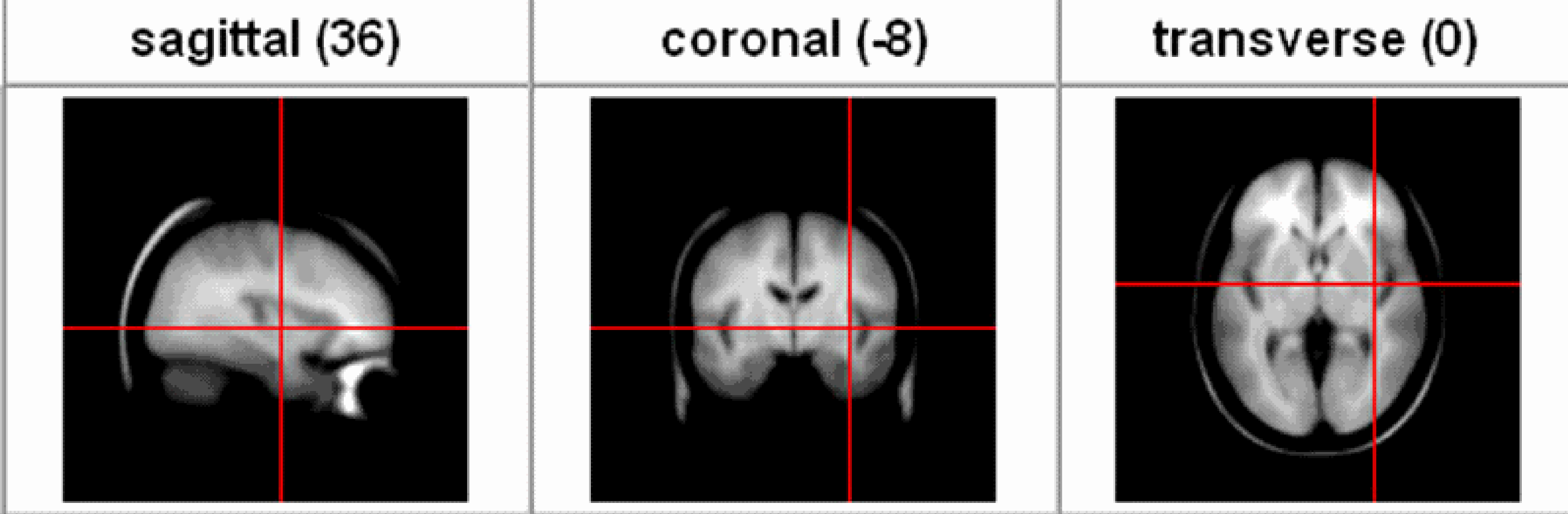
Figura 36: Imagem
do BrainWeb mostrando a média de intensidade de imagens de ressonância
magnética.
Devido a grande variabilidade presente no córtex cerebral, estas áreas apresentam-se demasiadamente borradas. O que limita muito seu uso como ferramenta de análise quantitativa.
Nos Atlas probabilísticos baseados em mapas de deformações, propriedades estatísticas destes mapas deformações são codificadas localmente para determinar a magnitude e tendência direcional da variação anatômica. A codificação da variação local pode então ser usada para abordar a severidade dos variantes estruturais fora da norma, os quais podem ser sinais de doença.
Atlas Baseado em Rótulos
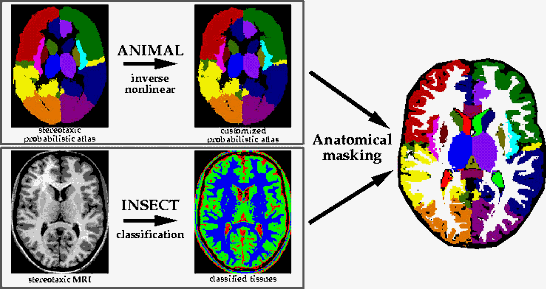
Figura 37: Mostra um método de rotulação automática com dois algoritmos usados em conjunto, o ANIMAL e o INSECT, para um Atlas probabilístico baseado em rótulos.
Segundo Toga et. al, as abordagens baseadas em rótulos, também chamadas de SPAM (Statistical/Probabilistic anatomy maps), grandes massas de dados cerebrais são manualmente segmentadas em sub-volumes, depois de mapear os conjuntos individuais no espaço estereotáxico.Um mapa probabilístico é então criado para cada estrutura segmentada, determinando a proporção dos rótulos de cada paciente em cada voxel no espaço estereotáxico.
Imagens de Cérebros, Atlas e afins
Atlas de Talairach Conjunto de slides de imagens de crio-secções de uma paciente alcoólatra de 60 anos, catalogadas e registradas em coordenadas estereotáxicas. O alinhamento do cérebro do paciente ao Atlas é feito de forma manual. O que torna a comparação ineficiente e dificulta a identificação de certas áreas sujeitas a grande variabilidade.
Áreas de Brodmann Mapa com informações cito-arquitetônicas que expressão áreas e suas funções. Sofre dos mesmos problemas do Atlas de Talairach.
Atlas de Schaltenbrand - Conjunto de slides de imagens de crio-secções de um cérebro marcado para evidenciar regiões talâmicas de um paciente, catalogadas e registradas em coordenadas estereotáxicas. Como o Atlas de Talairach, é necessário alinhar de forma manual e pode induzir ao erro.
Atlas de Matsui e Hirano - Conjunto de slides de imagens de crio-secções de alguns pacientes, catalogadas e registradas em coordenadas estereotáxicas.
Atamai Viewer e ASP (Atamai Surgical Planner) O ASP em conjunto com o Atamai Viewer compõe um pacote de visualização de MRI. Serve para fazer planejamento de cirurgias estereotáxicas. Mas usa um formato proprietário, o MNC.
ABA (Allen Brain Atlas) Atlas multi-modal que se destina ao estudo do cérebro do camundongo criando um mapa detalhado, de resolução a nível celular, que e englobe informação genética do cérebro do camundongo. O completo entendimento do cérebro do camundongo, mais simples que o cérebro humano, pode ajudar no entendimento futuro deste.
Cerefy Clinical Brain Atlas Atlas digital para planejamento de neurocirurgia baseado no Atlas de Schatenbrand e Talairach. Oferece uma plataforma integrada para o planejamento de cirurgia estereotáxica, e proporcionando uma base de comparação para identificação de tecidos saudáveis. O Cerefy é baseado no trabalho de Nowinski et. al. [23] de mapeamento cerebral e planejamento de cirurgias estereotáxicas.
BrainInfo Contém informação anatômicas sobre o cérebro de macacos, ratos e humanos. Sendo que a versão demonstrativa é bastante limitada. Serve como guia de referência para o estudo das estruturas cerebrais.
http://braininfo.rprc.washington.edu/
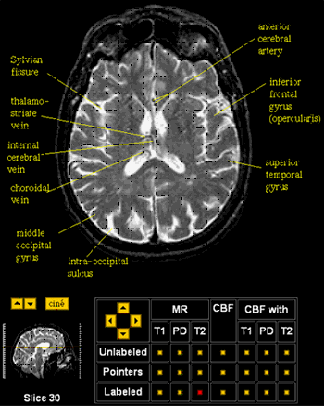
Figura 40: Demonstração do uso de BrainInfo localizando pela palavra chave "superior temporal gyrus" e selecionando o corte em MRI. Havia a possibilidade de escolher ver a dissecação do cérebro, mostrando a área escolhida na palavra chave; ou um diagrama da secção coronal do cérebro no modelo de Talairach; ou um mapa funcional. Também era possível escolher ver desenhos com linhas rotuladas em que aparecem a estrutura encontrado, mas esta função era para o cérebro de Macaca fascicularis.
The Whole Brain Atlas
(Universidade de Harvard) - Atlas probabilístico baseado em MRI.
Pode-se escolher navegar por um Atlas de um cérebro normal, ou visualizar
cérebros com determinadas doenças. Suas imagens podem ser
usadas para avaliar técnicas diversas aplicadas a MRI.
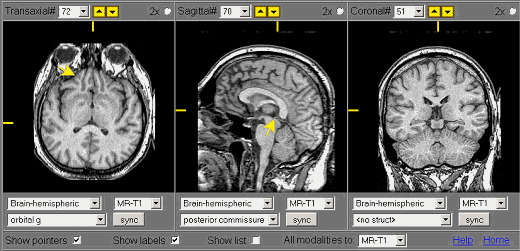
http://www.med.harvard.edu/AANLIB/home.html
Figura 41: Visualização de um série MRI de uma pessoa normal.
SPL/NSL Anatomy Browser Visualizador anatômico altamente detalhado e que usa um modelo hierárquico para catalogação da anatomia. Contém não só cérebros, mas é um Atlas anatômico de outras áreas do corpo também.
http://splweb.bwh.harvard.edu:8000/pages/papers/AnatomyBrowser/current/
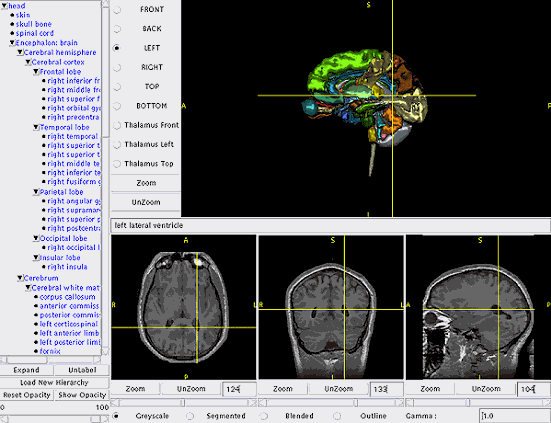
Figura 42: Mostra
uma tela de demonstração do Atlas anatômico do cérebro
no SPL/NSL Anatomy Browser.
Atlas of the Sheep Brain Atlas anatômico do cérebro da ovelha. Não muito útil, a não ser como guia de referência e como comparação anatômica com o cérebro humano.
Flybrain Atlas com informações anatômicas do cérebro da drosophila (Mosca).
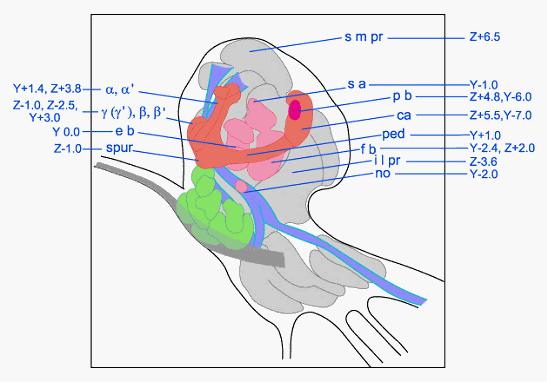
Figura 43: Esquema
do cérebro da drosophila em corte sagital.
BIRN SMART Atlas Atlas da BIRN (Biomedical Informatics Research Network), um consorcio entre várias universidades, que criou um Atlas do cérebro do camundongo usando GIS (Graphical Information Systems). O Atlas SMART (Spatial Markup and Rendering Tools) usa um sistema o sistema de coordenadas estereotáxicas Atlas do Cérebro do Camundongo de Paxinos e Franklin[18], tem seus marcos anatômicos derivados destes. Tem como propósito permitir que pesquisadores do campo da neurologia colaborar remotamente através de queries espaciais em imagens de cérebro.
http://www.geospatial-online.com/geospatialsolutions/article/articleDetail.jsp?id=130796

Figura 44: Esquema mostrando o funcionamento do BIRN SMART Atlas.
Digital Anatomist Project Contém informações anatômicas do cérebro reunidas em dissecações, reconstruções usando modelos tridimensionais, imagem de ressonância, enfim diversas modalidades. Serve ao estudo da neuroanatomia somente.
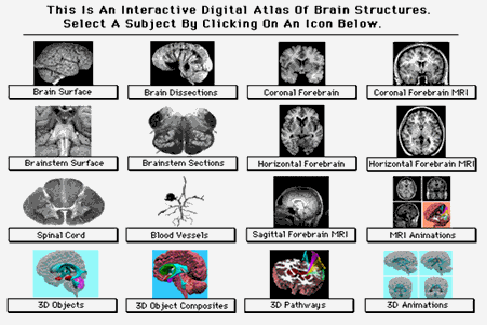
http://www9.biostr.washington.edu/da.html
Figura 45: Menu com as modalidades a serem escolhidas para se estudar o cérebro humano no Atlas do projeto Digital Anatomist.
MBL C57BL/6J Atlas Atlas multi-modal de um cérebro de camundongo C57BL/6J de 51 dias de idade.
PET Brain Atlas (PBA) O PET Brain Atlas é uma ferramenta on-line com o propósito educativo desenvolvido com para ensinar como interpretar diversos tipos de estudos de cérebros sob a modalidade PET.
Visible Human Project Conjunto de dados de Tomografias, Ressonâncias e Crio-secções de um indivíduo, compondo cerca de 15GB de informação. Os dados coletados também foram usados em outras iniciativas, inclusive uma sessão só da cabeça, mas nenhuma que o colocasse em um sistema de coordenadas estereotáxicas;
Comparative Mammalian Brain Collections Estudo comparativo entre diferentes mamíferos, organizados por sessões.
http://brainmuseum.org/
The Human Brain. Dissection of the Real Brain Atlas anatômico consistindo em dissecações do cérebro humano. Serve como guia de referência do cérebro e sistema nervoso associado.
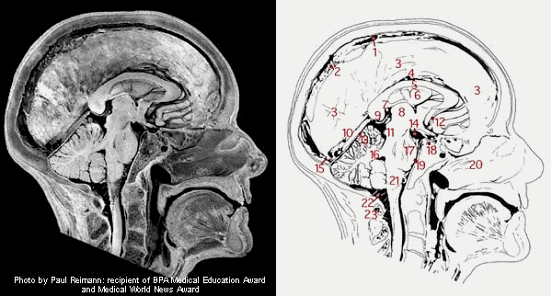
Figura 46: Corte anatômico
de uma cabeça. À direita um desenho com marcações.
LONI Human, Monkey, Rat and Mouse Atlases - (Laboratório de Neuroimagiamento da UCLA). Atlas bidimensional do cérebro de humanos, macacos, ratos e camundongos.
DTI Atlas Atlas de Imagiamento por Tensor de Difusão. Atlas Multimídia dos tratos da Substância Branca do Cérebro, usando DTI. DTI permite que se visualize a localização, orientação e anisotropia dos tratos da substância branca no cérebro, calculando a difusão das moléculas de água. Como mostra a figura abaixo. Observando os tratos cerebrais é possível evitar prejudicar uma determinada função cerebral caso seja necessário extirpar um tumor em uma cirurgia estereotáxica. Um tumor mais duro poderia apenas empurrar os tratos, e o cirurgião poderia poupá-los melhorando a chance de recuperação e de uma vida normal do paciente.
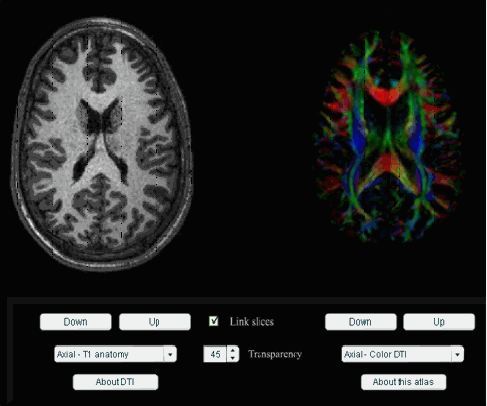
Figura 47: Mostra
um corte axial do cérebro humano na modalidade T1 à esquerda,
e à direita o rastreamento de fibras (fiber tracking) deste corte.
VOXEL-MAN Atlas anatômico
multi-modal com foco para o ensino de anatomia. Possui uma versão
somente com o cérebro e crânio.
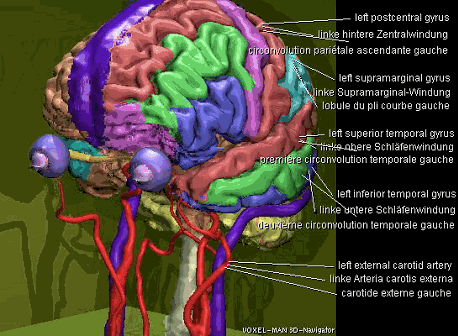
Figura 48: Tela de demonstração do Atlas Anatômico VOXEL-MAN.
4D-Probabilistic Atlas of the Human Brain Atlas probabilístico que pretende reunir dados clínicos, comportamentais, demográficos e de imagens de pelo menos 7000 indivíduos, de diversas etnias e coletados em diversas partes do globo. Até 2001 haviam sido coletados 5300 dos 7000, mas somente feito a triagem dos primeiros 500. Além dos dados supracitados, coletarão informações genéticas de 5800 pessoas. Das outras não será possível devido a restrições legais nos lugares de coleta. O Atlas ainda levará em conta não somente características anatômicas, mas também temporais levando em conta a idade dos indivíduos. Com este base de dados significativa, seria possível avaliar e comparar informações estatísticas de subpopulações sob uma abordagem uniforme, além de permitir o melhor estudo das similaridades e diferenças entre os indivíduos. Com efeito, 342 pares de gêmeos também fazem parte do conjunto de dados, de várias etnias e separados se desenvolveram ou não Alzheimer.
3.4 Ferramentas para Análise e Preprocessamento, Segmentação e Parcelamento
3D slicer programa para visualização, registro, segmentação e quantificação de dados médicos. Serve para orientar biopsias e craniotomias; oferecer visualização diagnóstica e planejamento cirúrgico; e facilitar a pesquisa em desvios cerebrais e estudos volumétricos.
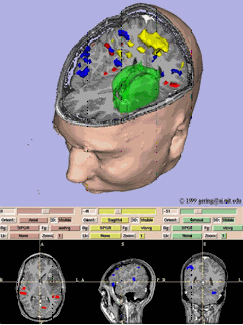
ABLe (Analyse of Brain Lesions) É um modulo do pacote MEDx para análise de lesões cerebrais.
AIR (Automated Image Registration) Ferramenta que permite o registro automático de iamgens 3D e 2D dentro do escopo do indivíduo ou entre vários indivíduos, ou dentro do escopo de uma modalidade ou, às vezes, entre modalidades diferentes.
http://bishopw.loni.ucla.edu/AIR5/index.html
ANIMAL (Automatic Nonlinear Image Matching and Anatomical Labeling) Algoritmo que deforma um volume MRI para que se encaixe em outro, previamente rotulado.
BrainMap Banco de dados de Neuroimagemento Funcional. Contém informação bibliográfica, descrição de experimentos e coordenadas tridimensionais de Talairach. Foi criada uma versão cadastrada chamada BrainMapDBJ, que eventualmente tornou-se oficial. http://www.brainmap.org
BrainVoyager O BrainVoyager
é uma pacote para análise e visualização de
de conjunto de dados de ressonâncias magnéticas funcionais
e estruturais.
BrainWeb (Montréal
Neurological Institute) Utiliza MRI scans de 305 jovens, para formar um
mapa da média voxel-por-voxel do volume estereotáxico dos
scans. Criando assim o Digital Brain Phantom.
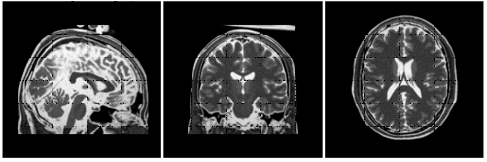
Figura 51: Demonstração do BrainWeb de um scan de modalidade T2 ICBM de uma pessoa normal. A espessura das chapas virtuais é de 1 mm o ruído calculado é de 3% e a não uniformidade da intensidade (RF) é de 20%, nesta série.
BRIAN (Brain Image Analysis) Um pacote para análise de imagens fMRI do cérebro humano. [24]
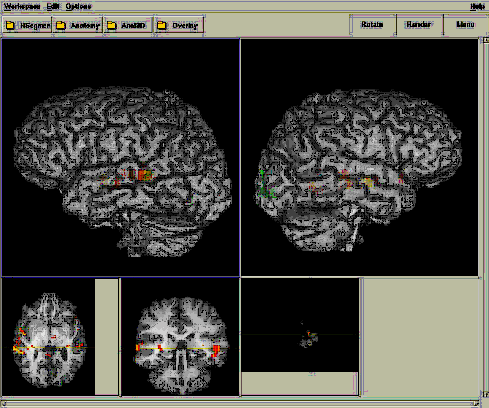
Figura 52: Uma das telas do BRIAN, uma reconstrução 3d e um registro 3d das áreas de ativação do cérebro em uma seqüência fMRI.
Display ferramenta de visualização e manipulação de dados tridimensionais do cérebro.
HAMMER Ferramenta para
registro elástico de imagens médicas, aplicados às
imagens de ressonância magnética do cérebro. Usa deformações
aplicadas a uma normalização espacial para melhor lidar com
variabilidades entre cérebros.
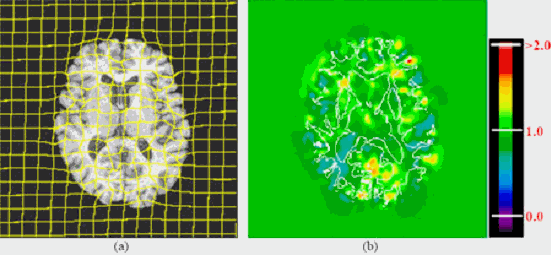
Figura 53: Transformações morfométricas efetuadas pelo HAMMER. Em (a) uma deformação em uma grade cartesiana e em (b) um mapa de cores do determinante do jacobiano da deformação em (a).
iBrain Pacote para análise e processamento de imagens de fMRI.
http://www.brain.org.au/software.html
INSECT (Intensity Normalized Stereotaxic Environment for the Classification of Tissue) É uma série de algoritmos usados para classificação de tecidos.
Lipsia Um pacote para análise de imagens fMRI do cérebro humano. Pode ser a continuação do BRIAN, pois é do mesmo laboratório. [25]
MEDx Pacote para análise e processamento de dados multimodais do cérebro. Usa o SPM para mapeamento estatístico e paramétrico do cérebro. É compatível com o padrão DICOM.
MindBoggle Uma abordagem
de espalhamento cerebral para rotulação automática.
Primeiramente separa-se a área da substância cinzenta do cérebro
e constrói a partir disto estruturas tridimensionais dos sulcos
dos cérebros do paciente e do Atlas previamente rotulado. [12]
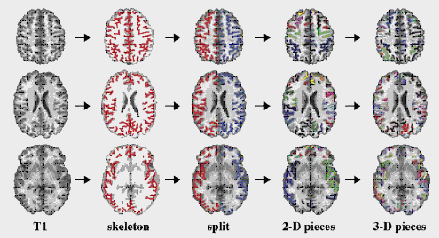
Figura 54:primeiro passo do processo MindBoggle de rotulação automática.
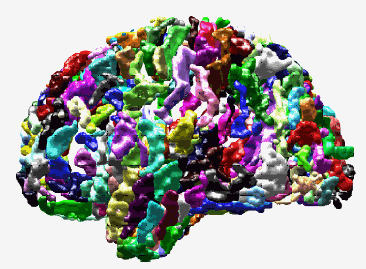 O
resultado será vários segmentos de objetos tridimensionais
espalhados. Como visto na imagem abaixo. As cores não representam
áreas específicas, mas foram escolhidas arbitrariamente.
O
resultado será vários segmentos de objetos tridimensionais
espalhados. Como visto na imagem abaixo. As cores não representam
áreas específicas, mas foram escolhidas arbitrariamente.
Figura 55: Modelos tridimensionais dos sulcos cerebrais após o processo de segmentação.
Depois cada pedaço é transformado e considerando o menor erro cada pedaço é alinhado ao do Atlas. As fronteiras são ajustadas, e os rótulos também se ajustam às novas fronteiras.
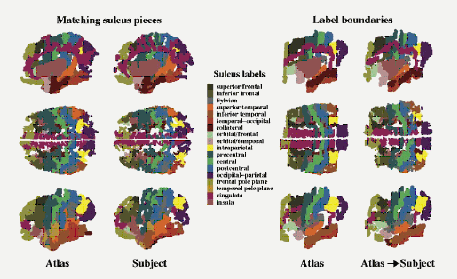
Figura 56: Alinhamento dos sulcos e transformação das fronteiras dos rótulos.
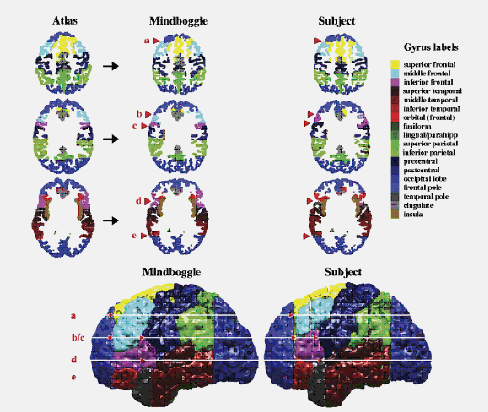
Figura 57: Último passo que é a transposição dos rótulos do Atlas para o cérebro do paciente.
SPM (Statistical Parametric Mapping) Refere-se à construção e abordagem de processos estatísticos de extensão especial usados para testar hipóteses sobre dados de imagiamento funcional. Projetado para análise de seqüências de dados de imagiamento cerebral. Última versão é a SPM2, mas a versão 5b está em desenvolvimento.
Talairach Daemon É o Atlas de Talairach em forma digital, usando um cliente Java.
http://www.loni.ucla.edu/ICBM/ICBM_Daemon.html
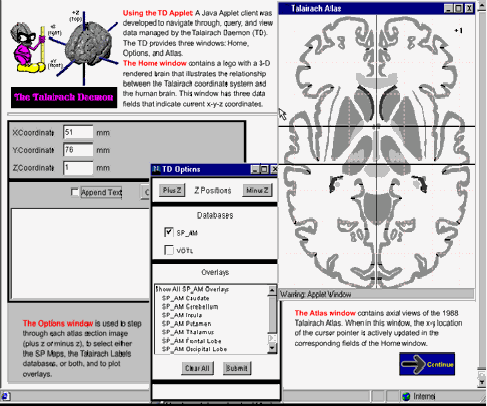
TSU (Talairach Space utility) Extensão para o SPM, que faz a visualização de Clusters do SPM99 em sessões do Atlas de Talairach.
3DVIEWNIX, Activ2000, AFNI, AMIDE, Anatomist, BAMM, BET, BRAINS2, Brain Tools, BSE, Caret, CCHIPS©, CirclePack, CLEAVE, Corner, DP Tools, EMMA, EvIdent (Fuzzy), FIASCO, fMRI Analysis Package (Yale), FMRISTAT, FMRLAB, FreeSurfer, FSL, GIFT, GpetView, LIMA, LOFA, Lyngby, MARINA, MIDAS (Tsui), MPITool, MRIcro, mri3dX, MRIVIEW, MRIWarp, MRVision,
N3, NeuroModeller, NPAIRS, PI-WAVE, PMOD, RPM, scanSTAT, SPAMALIZE, Stimulate, SUMA, SuMS, SureFit, Tina, UCLA BMD, VoxBo, WFU PickAtlas,
(X)MedCon
3.6 Fractais e o Córtex Cerebral, Alguma Relação?
O córtex cerebral humano é uma estrutura muito complexa. Possui certas características auto-similares e segundo Kiselev et. al. apesar das variabilidades anatômicas, na média, o córtex aparenta apresentar uma estrutura fractal.
As abordagens atuais recaem sobre a análise fractal do córtex cerebral. Diferentes estudos, como dito acima, usam desta abordagem para colher informações novas a cerca do cérebro. Usam de ferramentas matemáticas para análise fractal das estruturas a fim de reconhecer padrões de complexidade em indivíduos normais e em indivíduos que apresentam certa doença [5]. Com efeito, no trabalho de Thompson et. al [5], indivíduos com a síndrome de Williams apresentavam complexidade cortical maior, que em indivíduos normais.
No cérebro também a outros exemplos de fractais, como o padrão de veias e artérias e como elas se desenvolvem durante o crescimento do feto. E isto se repete em muitas outras partes do corpo. Fractais, então, tendem a ser a maneira com que a natureza constrói suas formas complexas e entender este processo pode facilitar a compreensão destas estruturas complexas. Estimativas recentes [13] apontam para um número de 100.000 genes codificados no DNA humano. Destes, cerca de 50.000, talvez, sejam ativos somente no cérebro e mostra a complexidade do controle genético sobre o cérebro e seu desenvolvimento.
Mas 50.000 é um número muito pequeno para englobar todas as sinapses, todas as posições de cada neurônio. Existem trilhões de sinapses no cérebro humano, diferente do cérebro da minhoca C. elegans a qual se mapeou completamente o cérebro. Na C. elegans todas as sinapses são codificadas pelos genes, e a falta de determinado gene faz com que a sinapse correspondente não se forme.
No homem, o processo de desenvolvimento do cérebro envolve influências genéticas além de outros fatores de desenvolvimento. Os mecanismos em si envolvem sinais químicos (onde cada célula teria uma afinidade com a outra e ligariam devido a esta afinidade química), competitividade celular (onde sinapses e células desnecessárias são removidas) e movimento guiado por fibras (onde neurônios são transportados a outras localidades através de fibras)[13].
O córtex cerebral possui uma estrutura em forma de camadas. O córtex visual, especificamente, possui várias colunas que processam os sinais vindos da retina em fases. As camadas são formadas de células que vão se multiplicando, param e formam uma camada, ou migram e se reposicionam para formar outras camadas.
Estes processos são observados em partes, mas não se entende como funcionam no geral, por que algumas células param de se reproduzir, por que outras migram. Mas como dito antes, não há como em 50.000 genes codificar todas as possíveis posições dos neurônios, o que sugere que exista uma base fractal neste processo.
O trabalho de Thomas Kromer [20] aborda essa suposição de uma outra forma. Usando fractais ele cria uma rede neural espacial, com dois algoritmos baseados no conjunto fractal de Julia, adaptado para gerar um sistema de coordenadas tridimensionais. Nessas redes neurais espaciais, as trajetórias das iterações são interpretadas como neurônios. E a iteração do algoritmo fractal leva de um ponto zn a um ponto zn+1.
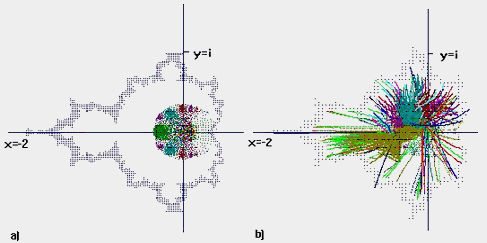
Figura 59: Mostra a comparação do conjunto de Mandelbrot, onde (a) forma uma estrutura parecida com o tálamo, e (b) os caminhos eferentes do "tálamo" à periferia que se assemelham conexões talâmicas reais.
A geração dessas redes neurais espaciais apresentou certas características que pareciam como algumas características presentes em cérebros. Como os conjuntos de Julia e Mandelbrot possuem propriedades de auto-similaridades e simetria nas estruturas geradas, a rede neural espacial gerada sobre essa premissa também as tem. Assim formaram-se estruturas parecidas com lobos, hemisférios, giros, sulcos e cavidades que se assemelham ventrículos.
Apresentar um modelo deformável adaptável ao cérebro específico de um paciente com exatidão usando um algoritmo de expansão fractal para o córtex cerebral humano. Para isto, criar um algoritmo em que se expandam, a partir de um modelo da estrutura basais do cérebro, os tecidos que compõe o córtex cerebral e, possivelmente, o córtex cerebelar. A fim de avaliar as vantagens dessa proposta em relação aos métodos atuais.
- Desenvolver um algoritmo de reconstrução tridimensional usando crescimento volumétrico fractal orientado por atratores colocados em marcos específicos da imagem de ressonância magnética, adaptável ao cérebro específico de cada paciente;
- Desenvolver uma ferramenta capaz de se adaptar as imagens geradas por qualquer equipamento, independente de serem ultramodernos ou mais antigos.
Características auto-similares do córtex cerebral podem prover outras pistas quanto à morfologia e ontogênese do córtex cerebral humano. Uma reconstrução tridimensional usando fractais poderia dar mais luz a essas pistas.
Os métodos atuais têm certas características que não levam se adaptam tão bem ao cérebro, principalmente ao córtex cerebral. Essa abordagem usando fractais visa suplantar algumas dessas dificuldades.
- Desenvolver um protótipo funcional que expanda um modelo tridimensional inicial usando características de crescimento fractal;
- Aperfeiçoar o protótipo para que a expansão assemelhe-se, ao final, um cérebro genérico;
- Testar o algoritmo para formas ideais e bem formadas de cérebros, visando identificar que marcos, e quantos, poder-se-ia usar para guiar o crescimento;
- Validar o algoritmo de renderização fractal em amostras diferentes de seqüências de imagens de cérebros;
- Comparar características entre as abordagens existentes e os resultados coletados.
- Mais facilidade na segmentação de regiões de interesse;
- Possibilidade de integração da análise fractal ao processo de visualização volumétrica do cérebro, e, com isso, dar outros recursos para detecção de patologias, mapeamento de funções, etc.
O trabalho limita-se a fazer a renderização somente, não se preocupando com a segmentação e rotulação das devidas áreas de acordo com um Atlas. Além disso, o trabalho não pretende recriar a ontogênese completa do cérebro do paciente, talvez apenas o que se refere ao estágio mais adulto do cérebro, na formação do córtex cerebral. A literatura [13] sugere envolver outros processos complexos e ainda não completamente entendidos que variam desde a formação do embrião e durante a vida adulta da pessoa.
Além disto, a o trabalho inicial limita-se criar um algoritmo de crescimento fractal que ao final assemelhe-se o cérebro do paciente, ou pelo menos, um cérebro normal. Abstendo-se da identificação de patologias ou análises mais aprofundadas das estruturas cerebrais. Visa identificar a viabilidade desta abordagem.
3.12 Referências Bibliográficas
[1] GLASER, Danya. Child Abuse and Neglect and the Brain. Environmental Influences on Brain Development. pág. 100-102. Acessado em: 5/5/2005, disponível em: http://www.ipce.info/library_3/files/glaser/glaser_2.htm
[2] TOGA, Arthur W. THOMPSON. Paul M. Maps of the Brain. The Anatomical Record. Vol 265. pag. 37-57. 2001.
[3] KISILEV, Valerij G. HAHN, Klaus R. AUER, Dorothee P. Is the brain cortex a fractal? Science Direct. NeuroImage Journal Vol. 20 pág. 1765-1774. 2003
[4] THOMPSON, Paul M. Bioinformatics and Brain Imaging: Recent Advances and Neuroscience Applications. Tutorial Chapter for Short Course in Bioinformatics, Williams, R.W. [ed.], Society for Neuroscience Conference, Orlando, FL, November 2-7, 2002. Acessado em: 23/03/2005, disponível em: http://www.loni.ucla.edu/~thompson/SFN2002/SFN2002_Course.html
[5] THOMPSON, Paul M. LEE, Agatha D. DUTTON, Rebecca A. GEAGA Jennifer A. HAYASHI, Kiralee M. BACHELLER, John D. ECKERT, Mark A. TOGA, Arthur W. REISS, Allan L. Fractal complexity of human cortex is increased in Williams Syndrome. International Conference of the Organization for Human Brain Mapping, Budapest, Hungary, June 13-17 2004. Acessado em: 3/5/2005, disponível em: http://www.loni.ucla.edu/~thompson/HBM2004/PT_HBM2004wms.pdf
[6] FREE, S. L. SISODIYA, S. M. COOK, M. J. FISH, D. R. SHORVON, S.D. Three-dimensional fractal analysis of the white matter surface from magnetic resonance images of the human brain. Cerebral Cortex, Vol 6. pag. 830-836. 1996.
[7] HOFMAN, M. The fractal geometry of convoluted brains. J. Hirnforschung. Vol 32. pag 103-111. 1991.
[8] TOGA, Arthur W. MAZZIOTTA, C. John. Brian Mapping The Methods. Ed. Academic Press. Canada, 1996
[9] CZANNER, S. DURIKOVIC R. INOUE, H. Growth Simulation of Human Embryo Brain. 2001. Acessado em: 17/11/2004, disponível em: http://www.cgg.ru/oct/dur/SCCG2001.htm
[10] KELEMEN, András. SZÉKELY, Gábor. GERIG, Guido. Elastic Model-Based Segmentation of 3-D Neuroradiological Data Sets. IEEE Transactions on Medical Imaging. Vol 18, número 10. Outubro de 1999. pag. 828-839.
[11] PITIOT, Alain. TOGA, Arthur W.. THOMPSON, Paul M. Adaptive Elastic Segmentation of the Brain MRI via Shape-Model-Guided Evolutionary Programming. IEEE Transactions on Medical Imaging. Vol 21, número 8. Agosto de 2002. pag.910-923.
[12] KLEIN, Arno. HIRSH, Joy. Mindboggle: a scatterbrained approach to automate brain labeling. NeuroImage, vol 24. 2005. pag. 261-280.
[13] THOMPSON, Richard F.. The Brain: A Neuroscience Primer. Chapter 10 Life Cycle of the Brain: Development, Plasticity and Aging. 3° Ed. Worth Publishers. Pag. 319-354. Nova York, Nova York. 2000.
[14] ROLAND, P.E. ZILES, K. 1994 Apud TOGA, Arthur W. THOMPSON, Paul M. Maps of the Brain. The Anatomical Record. Vol 265. pag. 41. 2001
[15] MAZZIOTA, John C.. et. al. 1995 Apud TOGA, Arthur W. THOMPSON, Paul M. Maps of the Brain. The Anatomical Record. Vol 265. pag. 41. 2001.
[16] NIELSEN, Finn Årup. Bibliography on Neuroinformatics. Neurobiology Research Unit, Rigshospitalet e Informatics and Mathematical Modeling, Technical University of Denmark. Abril de 2005. Acessado em 12/05/2005, diponível em: http://www2.imm.dtu.dk/~fn/bib/Nielsen2001BibNeuroinformatics/
[17] THOMPSON, Paul M. WOODS, Roger P. MEGA Michael S. TOGA, Arthur W. Mathematical/Computational Challenges in Creating Deformable and Probabilistic Atlases of the Human Brain. Human Brain Mapping, Vol 8. Pág. 81-92. 2000.
[18] PAXINOS, George. FRANKLIN, Keith B.J. The Mouse Brain in Stereotaxic Coordinates. Segunda Edição.
[19] FINNIS, Kirk W. STARREVELD, Yves P. PARRENT, Andrew G. SADIKOT, Abbas F. PERTES, Terry M. Three-Dimension Database of Subcortical Electrophysiology of Image-Guided Stereotactic Functional Neurosurgery. IEEE Transactions on Medical Imaging, Vol 22. Número1. Janeiro, 2003.
[20] KROMER, Thomas. Spatial Neural Networks Based on Fractal Algorithms Biomorph Nets of Nets of... Lecture Notes In Computer Science; Vol. 1625. Proceedings of the International Conference, 6th Fuzzy Days on Computational Intelligence, Theory and Applications. pag. 603 614. 1999
[21] REHM, Kelly. ROTTENBERG, David. SCHAPER, Kirt. STERN, Josh. HURDAL, Monica. SUMNERS, De Witt. Use of Cerebellar Landmarks To Define a Coordinate System and an Isolation Strategy. NeuroImage Human Brain Mapping 2000 Meeting. Poster número 536.
[22] FISHL, Bruce. SERENO, Martin I. TOOTELL, Roger B.H. DALE, Anders M. High-Resolution Intersubject Averaging and a Coordinate System for the Cortical Surface. Human Brain Mapping. Vol. 8. pag. 272-284. 1999.
[23] NOWINSKI, W.L. YANG, Guo Liang. YEO, Tseng Tsai. Computer-aided stereotactic functional neurosurgery enhanced by the use of the multiple brain atlas database. IEEE Transactions on Medical Imaging, Vol. 19, pag. 62-69. 2000.
[24] KRUGGEL, Frithjof. LOHMANN, Gabrielle. UHL, Christian. BRIAN (Brain Image Analysis) - Ein Programmsystem zur Analyse Multimodaler Datensätze des Gehirns. Max-Planck-Institut für neuropsychologische Forschung, Leipzig. Bildverarbeitung für die Medizin: Algorithmen, Systeme, Anwendungen, Proceedings des Aachener Workshops am 8. und 9. CEUR Workshop Proceedings , Vol 6. November 1996.
[25] LOHMANN, Gabrielle. MÜLLER, Karsten. BOSCH, Volker. MENTZEL, Heiko. HESSLER, Sven. CHEN, Lin. v. CRAMON, Yves. Lipsia - A New Software System for the Evaluation of Functional Magnetic Resonance Images of the Human Brain. Max Planck Institute of Cognitive Neuroscience e Max Planck Institute of Mathematics in the Sciences. Computerized Medical Imaging and Graphics, vol. 26, número 6. pág. 449-457. 2001.
NIELSEN, Finn Årup. Bibliography on NeuroInformatics. Neurobiology Research Unit, Rigshospitalet and Informatics and Mathematical Modelling, Technical University of Denmark. Abril de 2005. Acessado em: 07/05/2005, Disponível em: http://www2.imm.dtu.dk/~fn/bib/Nielsen2001Bib/
NIELSEN, Finn Årup. Bibliography on Segmentation on NeuroImaging. Neurobiology Research Unit, Rigshospitalet and Informatics and Mathematical Modelling, Technical University of Denmark. Abril de 2005. Acessado em: 07/05/2005, Disponível em: http://www2.imm.dtu.dk/~fn/bib/Nielsen2001Bib/
NIELSEN, Finn Årup.
Bibliography on Image Registration. Neurobiology Research Unit,
Rigshospitalet and Informatics and Mathematical Modelling, Technical University
of Denmark. Abril de 2005. Acessado em: 07/05/2005, Disponível em:
http://www2.imm.dtu.dk/~fn/bib/Nielsen2001Bib/



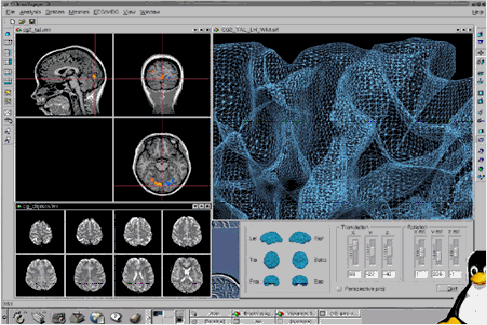 Figura
50: Tela da versão para Linux do BrainVoyager.
Figura
50: Tela da versão para Linux do BrainVoyager.